xLUNGS
Godna zaufania sztuczna inteligencja wspierająca identyfikację zmian chorobowych w płucach na bazie danych obrazowych
akronim: xLungs
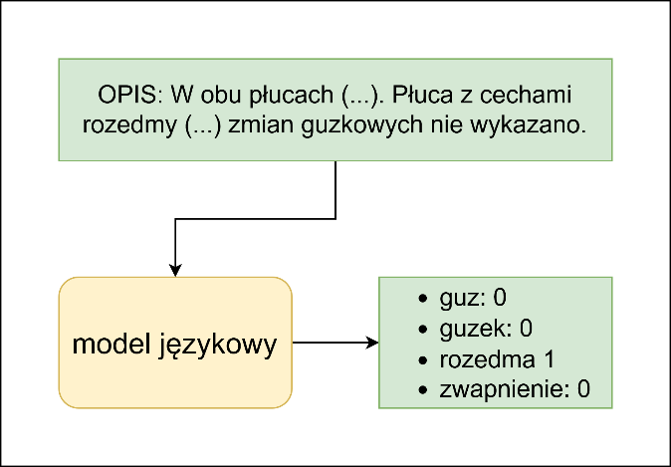
Celem projektu jest usprawnienie procesu identyfikacji zmian chorobowych, widocznych w badaniach TK i na zdjęciach rtg płuc. Cel ten realizujemy budując system informatyczny oparty o sztuczną inteligencję (SI) oraz interfejs użytkownika pozwalający radiologowi na konwersację z modułem SI. Narzędzie będzie zintegrowane z systemami gabinetowymi już wykorzystywanymi przez lekarzy.
Unikalną cechą proponowanego systemu jest moduł godnej zaufania sztucznej inteligencji, która:
1) skróci czas analizy zdjęcia potrzebny do wykrycia zmian chorobowych,
2) zapewni większą transparentność procesu oceny zdjęcia,
3) dostarczy wyjaśnień obrazowych oraz tekstowych wskazując przesłanki stojące za proponowaną rekomendacją,
4) będzie zweryfikowane pod kątem efektywności współpracy z radiologiem.
Projekt jest realizowany przez interdyscyplinarny zespół: specjalistów od inżynierii oprogramowania, sztucznej inteligencji, wyjaśnialnego uczenia maszynowego, wizualizacji danych oraz radiologii we współpracy z Polską Grupą Raka Płuca oraz Szpitalem Klinicznym w Białymstoku.
Okres realizacji projektu: 02.12.2021-31.05.2025
Wartość projektu: 5 755 000 zł
Kierownik projektu: prof. dr hab. inż. Przemysław Biecek
Projekt finansowany przez Narodowe Centrum Badań i Rozwoju w ramach konkursu INFOSTRATEG I


Z życia projektu

PvSTATEM
Testy serologiczne i leczenie P. Vivax: od randomizowanego badania klastrowego w Etiopii i na Madagaskarze do interwencji wspieranej technologią mobilną
akronim: PvSTATEM
Projekt PvSTATEM ma na celu wykazanie skuteczności i akceptowalności przez społeczność testów serologicznych i leczenia P. vivax (PvSeroTAT), nowej interwencji w zakresie kontroli i eliminacji malarii, w randomizowanych badaniach klastrowych w Etiopii i na Madagaskarze. Projekt wprowadzi również innowacje w zakresie nowych technologii mobilnych w celu skutecznego wdrożenia PvSeroTAT w warunkach wykraczających poza badania kliniczne. Interwencja PvSeroTAT obejmuje serologiczny test diagnostyczny, który mierzy przeciwciała przeciwko wielu antygenom P. vivax i informuje o indywidualnej decyzji dotyczącej leczenia. Jednak wyniki testów serologicznych mogą również informować o nadzorze nad malarią na poziomie populacji.
W tym projekcie Hop-on zostaną opracowane modele matematyczne, narzędzia uczenia maszynowego i technologie cyfrowe, aby dane generowane przez badania kliniczne w Etiopii i na Madagaskarze mogły informować o krajowych programach nadzoru malarii. Nasze konkretne cele to:
1) stworzenie oprogramowania komputerowego do przetwarzania danych diagnostycznych, które zostanie osadzone w prototypowych mobilnych aplikacjach zdrowotnych;
2) wprowadzenie innowacji w obecnych modelach matematycznych do analizy multipleksowych danych serologicznych generowanych przez test PvSeroTAT;
3) porównanie transmisji malarii na poziomie populacji w oparciu o serologiczne punkty końcowe ze standardowymi wskaźnikami transmisji malarii opartymi na częstości występowania pasożytów i częstości występowania klinicznego;
4) wykazanie przydatności i lepszej mocy statystycznej serologicznych punktów końcowych w badaniach klinicznych dotyczących kontroli malarii.
Okres realizacji projektu: 01.10.2020-30.09.2027
Budżet całościowy projektu: 6 794 427 EUR
Budżet PW w projekcie: 383 851,25 EUR
Kierownik projektu: prof. dr hab. inż. Przemysław Biecek
Z-ca kierownika projektu: dr Nuno Sepúlveda
Projekt realizowany przez interdyscyplinarne i uzupełniające się konsorcjum 9 partnerów z Europy, Afryki i Australii.
Projekt finansowany przez Unię Europejską w ramach programu ramowego Horyzont Europa (mechanizm Hop-on).



REFSA
Machine Learning-based systems for the automation of systematic literature reviews in food safety domain
akronim: REFSA
Celem projektu jest opracowanie systemu wspomagającego przeprowadzanie systematycznego przeglądu literatury (Systematic Literature Review – SLR) w obszarze bezpieczeństwa żywności.
Instytucje naukowe uczestniczące w ocenie ryzyka w obszarze bezpieczeństwa żywności mają obowiązek wydawania opinii dotyczących zanieczyszczeń chemicznych, dodatków do żywności, pozostałości pestycydów, nowej żywności i wielu innych zagadnień istotnych dla zdrowia publicznego. Przygotowanie opinii wymaga przeglądu dużej liczby prac naukowych (liczonych w tysiącach) związanych z badanym zagadnieniem. Istniejące obecnie aplikacje wspomagające SLR’y są mało przydatne w automatyzacji procesu poszukiwania pracy w obszarze bezpieczeństwa żywności, gdyż nie uwzględniają interdyscyplinarnego charakteru tej dziedziny wiedzy. System zaproponowany w projekcie jest oparty na multidyscyplinarnej wiedzy zawartej w ontologiach wykorzystywanych w tworzeniu reprezentacji numerycznych analizowanych artykułów. Na podstawie reprezentacji numerycznej artykułów dokonywana jest selekcja tych artykułów, które są przydatne przy formułowaniu opinii.
Okres realizacji projektu: 01.10.2020 – 01.01.2024
Całkowity budżet realizacji projektu: 5 983 369,13 zł
Wartość dofinansowania z budżetu państwa: 836 020,97 zł
Kierownik projektu: prof. dr hab. inż. Radosław Pytlak
Strona projektu: https://refsa-project.com
Projekt jest realizowany przez konsorcjum w składzie: Wydział MINI Politechniki Warszawskiej (lider konsorcjum), Narodowy Instytut Zdrowia Publicznego PZH – Państwowy Instytut Badawczy, Oslo Metropolitan University, Norwegian Scientific Committee for Food and Environment, Tecna, Sp. z o.o.
Projekt finansowany z funduszy norweskich w ramach programu „Badania stosowane”.


 Studies in English
Studies in English